NGMLR とSniffles
すっごく久しぶりになりますが、ロングリード用のマッピングツール(NGMLR)とその構造変異のツール(sniffles)に関する論文を少し細かく読んだので、ブログに書いておくことにしました。
Accurate detection of complex structural variations using single molecule sequencing
Nature Methodsに2018年4月に掲載されています(OAではありません)。
https://www.nature.com/articles/s41592-018-0001-7
プレプリントで同様の内容が読めます。
https://www.biorxiv.org/content/early/2017/07/28/169557
サプリメントがとても充実しているのと、方法の詳細は、サプリメントに記載があるため、詳細はサプリメントを中心に記載します。
この論文は、 Baylor College of MedicineのFritz Sedlazeck とUniversity of ViennaのPhillip Reschenederが開発しており、Fritz とPhillipは昔University of Viennaで研究をしていたようで、NGMLRを出す前にNexGenMap として論文をだしています。NGMLRはその発展版のような位置づけになるようです。
それぞれのツールの特徴を大きくまとめると以下。あ、このブログはおそらく長くなるので、ご容赦ください。
NGMLR
- Long Read 用に開発されたMapping ツール
- NextGenMap というMappingツールのLong Read 版
- NGMLRでは、アライメントのGap コストにConvex gap を利用している
Sniffles
- Deletion, Duplications, Insertions, Inversions, Translocations, Nested Events といった様々なタイプ(論文ではAll typesと記載)の構造変異の検出が可能
- Sniffles では、リード1本のアライメントに見つけることの出来る変異とSplit reads と呼ばれる場合の変異も検出ができる。
- Snifflesの新しい点は、新しいSVスコアリング方法。これによりエラーが多い、PacBioやONTのリードを使った構造変異検出に利用できるとしている。
ではNGMLRから
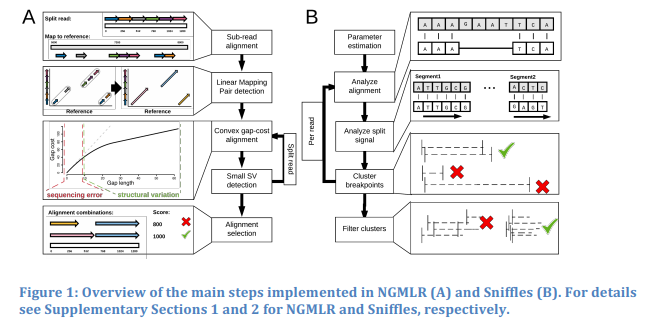
Fig. 1の図に大まかな2つのツールの流れが出ています。左がNGMLR、右がSnifflesですが、右のSnifflesは、さらに詳細な図が後述されるので、左図で、まずはNGMLRの説明をします。
Subsegment alignment とLMPの検出
まず、最初のステップがSubsegment alignment。図ではSub-readとなっていますが、のちに修正されているようなので、Subsegmentで行きます。
- Read を256bpにカット(Subsegment)し、Reference 配列へアライメント。
- Referenceスコアへ高いアライメントスコアでマップされた領域をSubsegementのマッピング領域候補とする。
- アライメントスコアが75%以下のものは捨てる
- 1000個以上マップされる場合(Repetitive領域など)は、
そのSubreadは捨てる。 - 残った候補から、Linear Mapping Pair (LMP) を定義.
- ゲノムに対して同方向で近い位置でマッピングされているペアを見つける
- このステップでは、SVを含んだ領域ではなく、まず精度よくマッピングされているサブセグメントの領域を見つけるというステージ。
サプリメントのFig 1.1が良いので入れておきます。
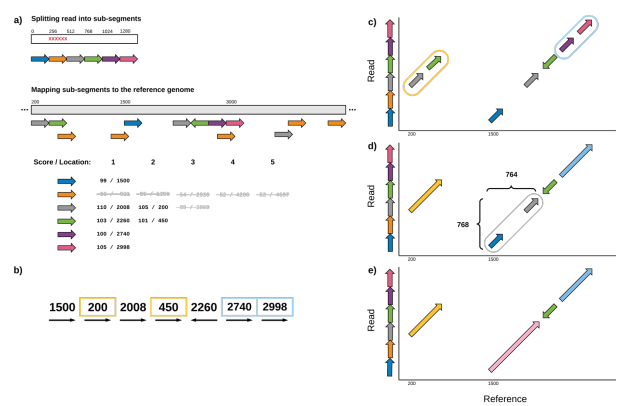
a)では、オーバーラップがないようにカットされ、 その候補が スコア/ポジション として記載されています。そうすると、1本のリードが複数個所にマップされ、そのうちいくつかポジションが近いものをペアとすることができます。b)の黄色の個所と青の個所です。これらはLMPとされます。そしてさらに、d)の2つのサブセグメントは、距離は離れてはいるものの離れた距離がReference上もリード上も同じ距離の場合、そのセグメントを結合してLMPとしています。
Convex Gap Alignment
そしてスコアの計算はConvex Gap スコア。ここでアライメントスコアのおさらいです(参照する配列と検索をかける配列の類似度を測るスコア)。
一般的なアライメントのGapスコア。上の配列を参照配列、下を検索をかけるクエリ配列とします。Wikipediaを参考にして記載しています ( Gap penalty - Wikipedia)。
Linear
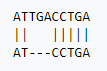
- Match score = 1, Gap cost = -1 の場合、上記アライメントは、7-(1)*3 = 4 となる
Affine
- Gap open cost, Gap extension cost を採用してスコアを計算。Gap open cost = -3, Gap extension = -1とすると前述のアライメントの場合は、7+(-3)+(-1)*3 = 1 となる
Convex
Affineと同様にGap Open cost を設定するが、Extension に定数×自然対数(Gapの長さ)というような設定をし、Gapが伸びた際のコストをマイルドにする。この論文では、もう少し違った式でConvex Gap を実装しています。
Gapスコアに関しては、構造変異が起きているような箇所は、小さくばらばらにGapが起こるのではなく、ある程度まとまったGap として観察されるという事を前提にGapのコスト計算がされています。
NGMLRでは、Convex を以下のように定義しています。

g_o は、Gap Open コストでGap を開くときのコスト、g_e はExtension コストで、gapを伸長するときにかかるコスト、g_MはGapペナルティが大きく0以下にならないようにするMinimumの値、そしておそらくこのDecay を入れているのが新しいのかなと思いますが、g_D として定義しているGap Decay コスト。
少し細かくなりましたが、サプリメントのFig 1.2 に良い図があります。
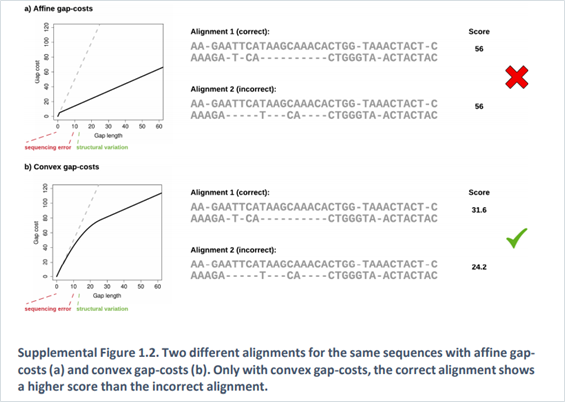
著者らが実装したNGMLRのConvexでは、大きなGapを受け入れやすくなっています。Fig 1.2 でも、Affineコストでは、大きなGapと複数のGapが同じスコアになるのに対して(a)、採用したConvexでは、大きなGapがより大きいスコアとなっていることからもわかります(b)。
Small InDel Detection
次のステップは、小さなInDelの検出です。
小さなSmall Inversionの検出のために以下を行っています:
- LMPの3bpごとのWindowsでアライメントスコアを計算
- スコアがある閾値(65%)より小さいものがある場合、その領域を抜き出す
- Reverse Compliment を作成し、それがReferenceにマッチし、アライメントスコアが上がるか調べる
これもサプリメントに良い図があるので、記載しておきます。
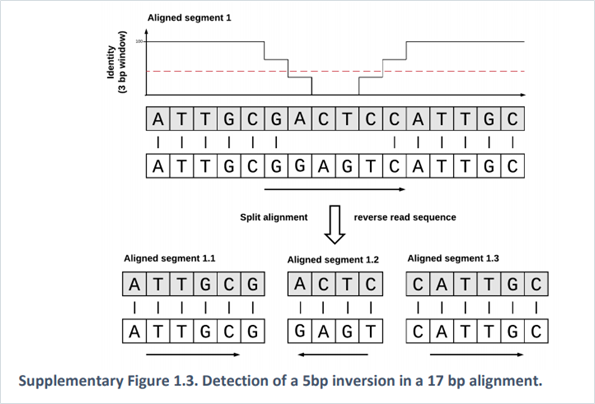
Alignment Selection
NGMLRで最後のステップは、Alignment Selection です。LMPでは、1つのリードが複数個所に分断されてマップされる個所を見つけていますが、ゲノムビューアで閲覧できるように、1つのリードをどのポジションにマップさせるか、決めなくてはいけません。そのために以下のようにしてマップさせるポジションとマッピングクオリティを決めています。
- Linear Mapping Pair (LMP)の組み合わせで、オーバーラップなく、もっともスコアが高くなる組み合わせを選択
- Mapping Quality は、SubreadsのMapping Qualityの平均
そしてようやく次がSnifflesです。
これもサプリメントに詳細なプロセス図があるので、まずはそちらを掲載します。
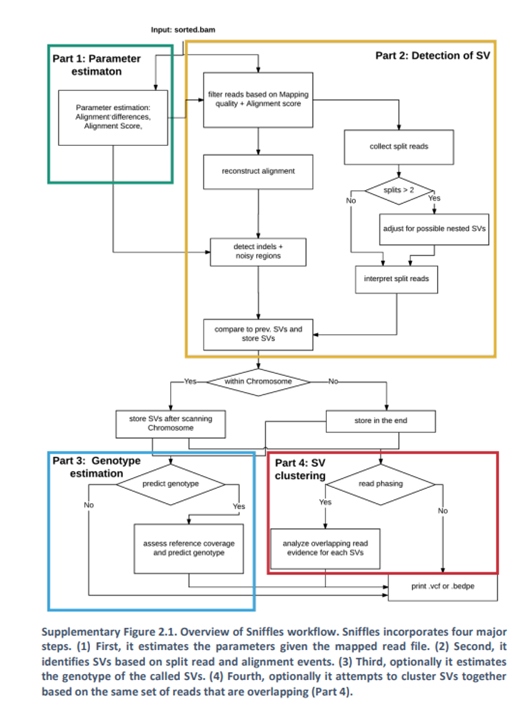
Prameter Estimation
最初のステップは、パラメータの推定です。Snifflesでは、3つのパラメータを推定します。このパラメータ推定は、ランダムに選択した10,000本のインプットリードから行います。SVは稀に観察される事象であるという仮定で、この10,000本がSVにオーバーラップせずにベースの値として利用できることを想定しています。
- アライメント中のIndelやMismatchによる違い。
- 100塩基中にみられるのミスマッチとIndelの個数の95%分位点。
- 各リードがマップされたBestと2番目に良かった場所のアライメントスコアの比
Detection of SV
Filtering : 以下の条件に当てはまるものは捨てる
- Mapping Quality < 20
- Bestと2番目のアライメントスコアの比が2以下 (つまり似ている箇所のアライメントがある)
- 7か所以上にSplitしたリードがマップされる場合
そして次のステップでSVの領域を検出します。
- まず最初に小さな(<1kb)Insertion, Deletion とNoisyな領域(ミスマッチが多く、1-5bpのInDelが見つかる領域)の検出。
-
1つのアライメントでは説明できない大きなGap(Split reads)によるSVを検出
Split reads の情報は、マッピング結果のSAM/BAMファイルのSAタグを利用
- ステップ1と2で検出されたSVを支持するリードは、それらが同じSVに由来するか分類
- 疑わしいSVは捨てる
サプリメントのFig 2.4 にSnifflesが検出できるSVと、Split reads のマップのされ方が記載されています。
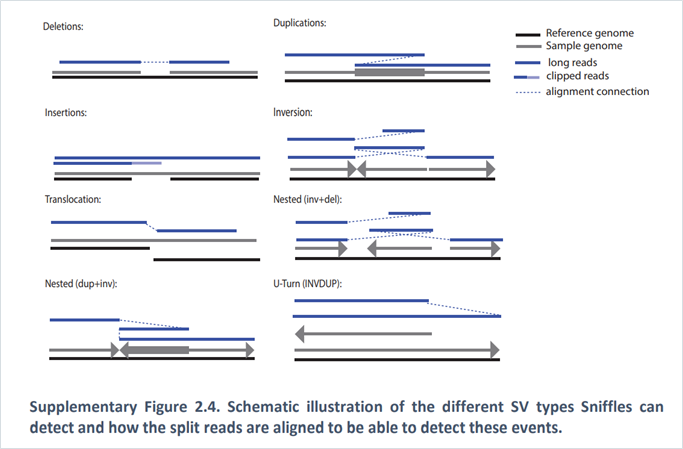
SVをそれぞれ上記分類に分けるプロセスは以下。
1.異なるアライメントが同じ染色体で同方向の場合、かつ長さが指定したSVの長さ(default = 30bp)っよりも長い場合、Insertion とコール。Deletion も同様にコール。Duplicationは、Reference上の距離がユーザーが指定するSVの距離よりも大きく、またリード側の距離がユーザーが指定するSVの距離より小さい場合にDuplicationとコール。
2.2つのセグメントが同じ染色体上にあるが、異なるStrandにある場合、Inversion とコール。2つのセグメントが少なくとも200bpオーバーラップし、長さが40%以上すくないものはInversion Duplicatin (U-Turn)とコール
3.2つのセグメントが異なる染色体にある場合は、Translocation とコール
Break pointの検出
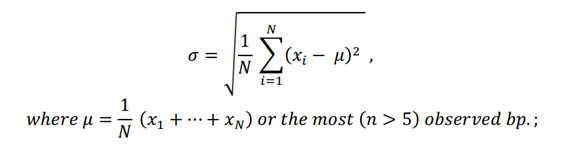
- 1st、4th quantile (5%, 95%)のばらつきを持つリードを除去することで、より正確なBreak point を検出できるとしている。
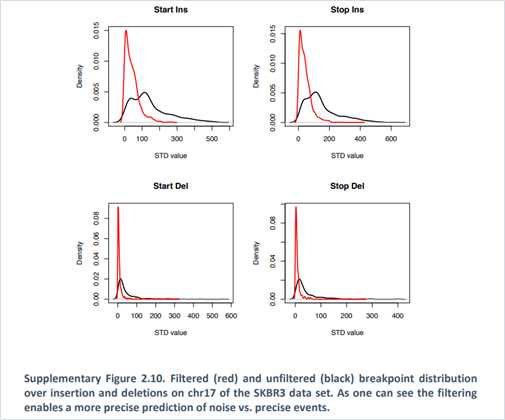
と、こんな感じが、SVの検出のところまでです。Snifflesはこのほか、ジェノタイプの検出やフェージングも行えるようになっています。
このツールを使った結果は、シミュレーションと実データでの検出が行わわれていますが、その辺りは論文に分かりやすく記載されていると思います。
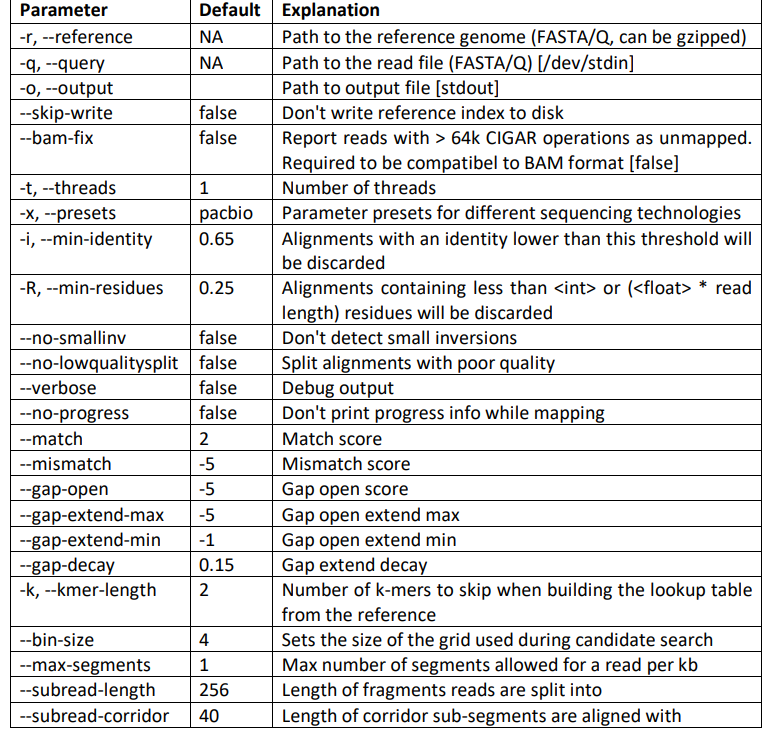
補足としてパラメータの記載がサプリメントに会ったので、入れておきます。
NGMLRのパラメータ
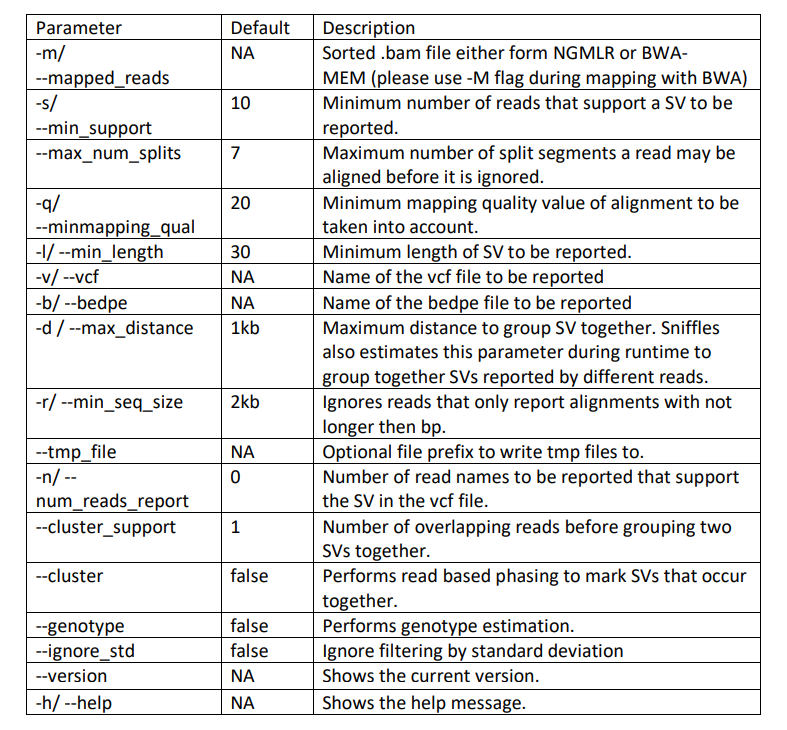
Snifflesのパラメータ
以上になります。また時々こういうブログが書けたらいいなと思います。お気づきの点がありましたら、ご連絡ください。